 Supongo que todos los fans de Natalie Portman saben que firmó dos artículos científicos como Natalie Hershlag en 1998 y en 2002, pero yo me acabo de enterar gracias a un tuit de Alejandro Rivero (@arivero). En secundaria fue primera autora de un artículo publicado en la revista Journal of Chemical Education (enviado al Intel Science Talent Search). En 1999 se matriculó en Psicología en la Universidad de Harvard, publicando un segundo artículo con cinco coautores en la revista NeuroImage; acabó la carrera (bachelor) en 2003. Todo ello entre el rodaje de los episodios II y III de Star Wars y otras películas. Muchos de sus fans pueden utilizarla como ejemplo a imitar, una buena estudiante que invierte parte de su tiempo en aprender a investigar. No sé, ahora veo con otros ojos a Portman. Los artículos son Natalie Hershlag, Ian Hurley, Jonathan Woodward, «A Simple Method To Demonstrate the Enzymatic Production of Hydrogen from Sugar,» J. Chem. Educ. 75: 1270–1274, 1998 [pdf gratis], y Abigail A. Baird, Jerome Kagan, Thomas Gaudette, Kathryn A. Walz, Natalie Hershlag, David A. Boas, «Frontal Lobe Activation during Object Permanence: Data from Near-Infrared Spectroscopy,» NeuroImage 16: 1120–1126, 2002 [pdf gratis]. Sigue leyendo
Supongo que todos los fans de Natalie Portman saben que firmó dos artículos científicos como Natalie Hershlag en 1998 y en 2002, pero yo me acabo de enterar gracias a un tuit de Alejandro Rivero (@arivero). En secundaria fue primera autora de un artículo publicado en la revista Journal of Chemical Education (enviado al Intel Science Talent Search). En 1999 se matriculó en Psicología en la Universidad de Harvard, publicando un segundo artículo con cinco coautores en la revista NeuroImage; acabó la carrera (bachelor) en 2003. Todo ello entre el rodaje de los episodios II y III de Star Wars y otras películas. Muchos de sus fans pueden utilizarla como ejemplo a imitar, una buena estudiante que invierte parte de su tiempo en aprender a investigar. No sé, ahora veo con otros ojos a Portman. Los artículos son Natalie Hershlag, Ian Hurley, Jonathan Woodward, «A Simple Method To Demonstrate the Enzymatic Production of Hydrogen from Sugar,» J. Chem. Educ. 75: 1270–1274, 1998 [pdf gratis], y Abigail A. Baird, Jerome Kagan, Thomas Gaudette, Kathryn A. Walz, Natalie Hershlag, David A. Boas, «Frontal Lobe Activation during Object Permanence: Data from Near-Infrared Spectroscopy,» NeuroImage 16: 1120–1126, 2002 [pdf gratis]. Sigue leyendo
Archivo de la etiqueta: Bioquímica
Francis en Eureka: Por qué los españoles tienen una piel clara
Puedes escucharme en ¡Eureka!, mi sección en el programa La Rosa de los Vientos, en Onda Cero, hablando de despigmentación de la piel y síntesis de la vitamina D. Como siempre una transcripción, enlaces a los artículos técnicos y noticias, e imágenes.
La especie humana nació en África, donde el color de la piel predominante es oscuro, y emigró hacia Europa, donde predomina el color claro. ¿Qué ventaja tiene el color claro de la piel en Europa? La gran ventaja es la síntesis de la vitamina D necesaria para el crecimiento, la correcta mineralización de los huesos y el desarrollo del esqueleto. La vitamina D tiene una estructura química muy parecida a la del colesterol. Hay varios tipos, pero los más importantes son la vitamina D3, colecalciferol, que es la forma de la vitamina D que se encuentra en los animales, y la vitamina D2, ergocalciferol, que es la forma que aparece en las plantas. Hay pocos alimentos vegetales o animales que contengan vitamina D, pero contienen precursores inactivos, llamados provitaminas, que necesitan radiación ultravioleta para su activación no enzimática. Por eso para sintetizar vitamina D tiene que darnos el sol sobre la piel.
Pero la radiación ultravioleta es peligrosa para la piel y cuando vamos a la playa tenemos que usar algún tipo de protector solar para evitar la aparición de melanomas. Los melanomas son tumores pigmentados que aparecen cuando nos exponemos mucho al sol y pueden producir ciertos tipos de cáncer de piel. El color de la epidermis se debe a la melanina presente en los melanocitos y sirve de protección contra la radiación ultravioleta. En las zonas intertropicales hay tanta irradiación solar que predominan las pigmentaciones oscuras de la piel, sin que ello afecte a la síntesis de la vitamina D. Pero en Europa la evolución ha tenido que seleccionar entre prevenir el melanoma, que es una enfermedad que aparece en la época postreproductiva, o favorecer un correcto crecimiento de los huesos en la época prerreproductiva. Por lo que parece la evolución ha favorecido la despigmentación a costa de un mayor riesgo a padecer melanoma.
El artículo técnico es Conrado Martínez-Cadenas, Saioa López, et al. «Simultaneous Purifying Selection on the Ancestral MC1R Allele and Positive Selection on the Melanoma-Risk Allele V60L in South Europeans,» Molecular Biology & Evolution, AOP Sep 17, 2013. Llamó mi atención sobre este artículo la noticia de Laura M. Zahn, «Sunny Days,» Editor’s Chooice, Science 342: 163, 11 Oct 2013, y también «¿Por qué en el sur de Europa tenemos la piel clara?,» UPV/EHU, Agencia SINC, 04 oct 2013.
Nobel Química 2013: Karplus, Levitt y Warshel por la bioquímica computacional
El austríaco Martin Karplus (Univ. Harvard, Cambridge, Massachusetts, EEUU), el sudafricano Michael Levitt (Facultad de Medicina de la Univ. Stanford, California, EEUU) y el israelí Arieh Warshel (Univ. del Sur de California, EEUU) son los ganadores del Premio Nobel de Química 2013 por «el desarrollo de modelos multiescala para sistemas químicos complejos.» La simulación por ordenador de la química cuántica de las macromoléculas (como las proteínas) y sus interacciones con los metabolitos (moléculas pequeñas) es imposible; el número de grados de libertad crece de forma exponencial con el tamaño. Por fortuna, basta simular la física cuántica de la reacción en el sitio activo (o centro de reacción), pudiendo usarse la mecánica clásica de Newton para simular las vibraciones del resto de la molécula, la llamada dinámica molecular. Este tipo de simulación multiescala fue introducida por Karplus, Warshel y Levitt entre 1972 y 1976. Desde entonces se considera la técnica numérica estándar para simular procesos bioquímicos en macromoléculas.
Anuncio del premio Nobel, información divulgativa [PDF], información técnica [PDF] y un artículo periodístico de Antonio Martínez Ron, «Nobel de Química 2013 para los científicos que facilitaron las simulaciones químicas por ordenador,» lainformacion.com, 9 Oct 2013.
El coste enzimático de la eficiencia en el metabolismo energético
Todo el mundo sabe que el coste de una obra de teatro además del trabajo de los actores incluye a mucho personal entre bambalinas. El coste termodinámico de una ruta metabólica también incluye un coste entre bambalinas, el coste proteómico de la síntesis de sus enzimas. Nos lo recuerda un nuevo artículo en PNAS que estudia en procariotas la ruta metabólica de la glucólisis, la síntesis de energía (moléculas de ATP) oxidando glucosa. La ruta metabólica estándar para la glucólisis es la de Embden-Meyerhoff-Parnass (EMP), que contiene 10 reacciones enzimáticas y cuyo producto son dos moléculas de ATP. Sin embargo, hay otras rutas para la glucólisis en procariotas, siendo la más común la ruta de Entner-Doudoroff (ED), que produce una sola molécula de ATP, pero sólo tiene 6 reacciones enzimáticas. Avi Flamholz (Instituto Weizmann de Ciencia, Rehovot, Israel) y sus colegas afirman que la prevalencia de la ruta ED en procariotas es debido a que requiere menos enzimas (6 en lugar de 10); este resultado lo han obtenido tras un análisis termodinámico de la cinética química de estas rutas metabólicas que incluye el coste termodinámico de la producción de las enzimas (normalmente despreciado en este tipo de análisis). El nuevo estudio nos recuerda que las bambalinas también existen en el metabolismo celular. Nos lo cuentan Arion I. Stettner, Daniel Segrè, «The cost of efficiency in energy metabolism,» PNAS, AOP May 31, 2013, quienes se hacen eco del artículo técnico de Avi Flamholz et al., «Glycolytic strategy as a tradeoff between energy yield and protein cost,»PNAS, AOP Apr 29, 2013.
Fotografian un haz de siete moléculas de ADN suspendido entre dos micropivotes
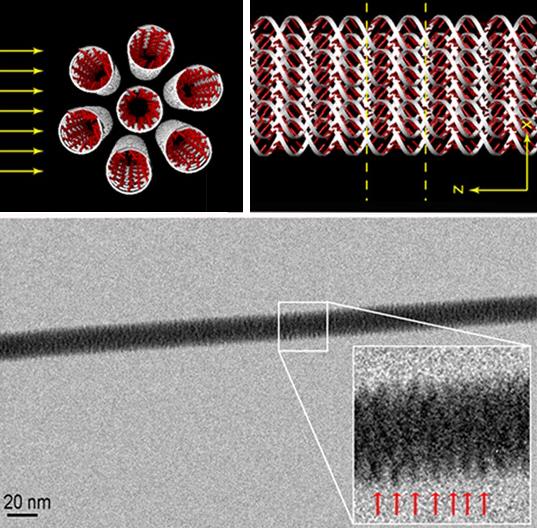
Esta fotografía obtenida con un microscopio electrónico de transmisión (TEM) muestra una fibra formada por un haz de siete moléculas de ADN en una configuración 1+6, un ADN central rodeado de 6 ADN en una distribución hexagonal; todos tienen la configuración ds ADN lineal (ds λ-DNA config. A). Para lograr esta fotografía electrónica han suspendido el haz de moléculas de ADN entre dos micropivotes de silicio y han hecho un agujero entre ellos por el que ha penetrado el haz de electrones del microscopio. Todo un alarde técnico para una foto que dejará frío a muchos, acostumbrados a ver reconstrucciones 3D de la molécula de ADN átomo a átomo. En esta fotografía la molécula de ADN se intuye (no me atrevo a decir que se ve) en los lugares donde apuntan las flechitas rojas, colocadas para ilustrar la periodicidad del enrollamiento de una de las moléculas de ADN; las flechitas rojas están separadas por 2,7 ± 0,2 nm, cada molécula de ADN enrollada tiene un diámetro de unos 8 nm y el haz 1+6 moléculas de ADN tiene unos 19 nm. El artículo técnico es Francesco Gentile et al., «Direct Imaging of DNA Fibers: The Visage of Double Helix,» Nano Letters, ASAP Nov. 22, 2012.
Unas sencillas reglas permiten obtener la conformación terciaria de una proteína a partir de la secundaria
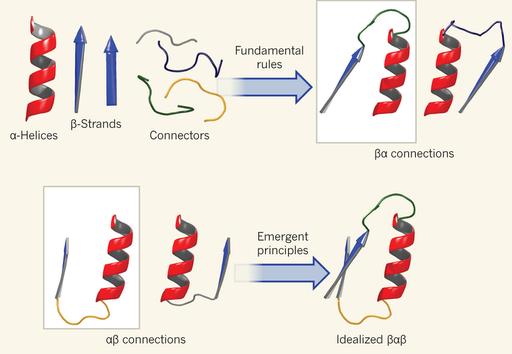
Uno de los problemas más importantes del s. XXI es el problema del plegamiento de proteínas, determinar la estructura tridimensional (conformación terciaria) de una proteína a partir del listado de sus aminoácidos (o de su código genético). Esta estructura nativa es única para la mayoría de las proteínas, determinando en gran parte su función bioquímica (la geometría determina la función). Se ha publicado en Nature un artículo que propone los principios básicos y las reglas fundamentales que controlan el plegamiento a partir de la estructura secundaria de las proteínas (las hélices α y las hojas β). Estos principios podrían usarse para diseñar nuevas proteínas que se plieguen de la forma deseada, lo que podría tener enormes aplicaciones en biología sintética. Nos lo cuenta Birte Höcker, «Structural biology: A toolbox for protein design,» Nature 491: 204–205, 08 November 2012, que se hace eco de Nobuyasu Koga, Rie Tatsumi-Koga, Gaohua Liu, Rong Xiao, Thomas B. Acton, Gaetano T. Montelione, David Baker, «Principles for designing ideal protein structures,» Nature 491: 222–227, 08 November 2012.
El dilema del prisionero, la cooperación molecular y el origen de la vida

El dilema del prisionero de Tucker, un juego de suma no nula, demuestra que la no cooperación, el egoísmo puro y duro, resultado del equilibrio de Nash, puede ser la mejor solución de muchos problemas. Hay variantes del juego con la conclusión opuesta, en las que la cooperación es la solución de equilibrio. Hay moléculas orgánicas por doquier en el universo, sin embargo, el origen de la vida en la Tierra sigue siendo aún un gran misterio. Cómo un conjunto de moléculas orgánicas inanimadas se autoorganizó para dar lugar al primer ser vivo. Nilesh Vaidya (Universidad Estatal de Portland, Oregon, EEUU) y sus colegas sugieren en Nature que la cooperación entre las moléculas pudo contribuir al origen de la vida. En cierto sentido, estas ideas son opuestas a la teoría del gen egoísta de Richard Dawkins, ya que el egoísmo, igual que en el dilema del prisionero, no conduce a la solución óptima. El nuevo artículo muestra ejemplos de redes de moléculas de ARN que se ensamblan entre sí gracias a la cooperación molecular, lo que sugiere que ésta puede ser tan antigua como la vida misma. Como es obvio, sus ideas se enmarcan dentro de la hipótesis del «mundo de ARN» que sugiere que la biología primordial carecía de ADN y de proteínas, siendo el ARN responsable tanto de la herencia como del metabolismo. El nuevo artículo es realmente sugerente, aunque todavía queda mucho para que resolvamos el misterio del origen de la vida. Nos lo cuentan James Attwater & Philipp Holliger, «Origins of life: The cooperative gene,» Nature, Published online 17 October 2012, que se hacen eco del artículo técnico de Nilesh Vaidya, Michael L. Manapat, Irene A. Chen, Ramon Xulvi-Brunet, Eric J. Hayden & Niles Lehman, «Spontaneous network formation among cooperative RNA replicators,» Nature, Published online 17 October 2012 (recomiendo a los biomatemáticos echar una ojeada al modelo matemático que aparece en la Información Suplementaria del artículo).
Premio Nobel de Química 2012: R. J. Lefkowitz y B. K. Kobilka por los receptores acoplados a las proteínas G y su función

Las células eucariotas perciben moléculas de su entorno gracias a una serie de proteínas que se encuentran en su membrana, las más importantes son los receptores acoplados a proteínas G (los GPCR, siglas de G-protein-coupled receptors); las proteínas G son las que inician la cadena de señalización celular asociada a dichas moléculas y ya fueron objeto del Premio Nobel de Medicina en 1994, concedido a Alfred G. Gilman y Martin Rodbell. Estos receptores se ligan a compuestos fotosensibles, olores, feromonas, hormonas y neurotransmisores, activándose y permitiendo que a ellos se liguen las proteínas G correspondientes. Casi la mitad de los medicamentos modernos utilizan estos receptores como diana pues los GPCR están involucrados en muchas enfermedades. En 1968, Robert J. Lefkowitz (ahora en la Univ. Duke, Durham, Carolina del Norte, EEUU) usó isótopos radioactivos en la hormona adrenalina para trazar los receptores transmembrana de las células que se acoplaban a ella; en concreto, descubrió el receptor adrenérgico β de la adrenalina, que publicó en 1970 en PNAS y Science. En los 1980, Lefkowitz decidió estudiar los genes asociados a las GPCR y contrató a un joven doctor, Brian K. Kobilka (ahora en la Univ. Stanford, California, EEUU) que aisló el gen que codifica el receptor adrenérgico β y descubrió que es similar a uno que captura luz en el ojo (el momento Eureka de este Premio Nobel). Gracias a ello descubrió una nueva familia de receptores cuya estructura molecular y función es similar, los GPCR, que entonces estaba constituida por unos 30, pero que hoy en día comprende unos mil receptores. El motivo de la concesión del Nobel de Química de este año a estos dos investigadores ha sido uno de las noticias de 2011, la publicación en Nature por Kobilka del mecanismo exacto de funcionamiento del receptor adrenérgico β, gracias a imágenes de este receptor en su estado activado con la adrenalina. Una imagen que culmina varias décadas de investigación. Nota de prensa, información para un público general, información técnica y anuncio del premio.

Esta figura muestra el funcionamiento de los GPCR, que se activan (1) al ligarse a una hormona (por ejemplo); en su forma activa (2) se ligan a las proteínas G que se disgregan en una subunidad alfa (3) que inicia una cadena de señalización celular, que altera el metabolismo celular (el efecto de la hormona); el proceso se repite de nuevo muchas veces (4). La clave de este proceso son los cambios estructurales que se producen en los GPCR cuando se activan, que fue desvelado en 2011 por el grupo de Kobilka que logró obtener una imagen cristalográfica de rayos X de un GPCR activado, en concreto, el receptor adrenérgico β de la adrenalina. La imagen para dicho GPCR sin activar ya era conocida desde hacía mucho tiempo, pero cristalizar un GPCR activado requirió dos décadas de trabajo (mucha gente pensaba que era imposible) y mereció un artículo en Nature en 2011, la clave para la concesión este año del Premio Nobel.

Gracias a la determinación de la estructura tridimensional (o conformación) de un GPCR activado se ha podido elucidar cómo funciona a nivel bioquímico; como la mayoría de los GPCR comparten gran parte de su estructura, este gran avance permitirá diseñar mejores fármacos agonistas (moléculas capaces de combinarse con un receptor y estimular su actividad), antagonistas (moléculas capaces de bloquear un receptor y abolir su actividad) y agonistas inversos (los que logran efectos opuestos a los de los agonistas). Los agonistas se ligan a un GPCR y estabilizan la conformación que activa la proteína G en el interior celular. Los agonistas inversos se ligan a un GPR pero estabilizan su conformación no activada. Los antagonistas o inhibidores compiten con los agonistas y bloquean el sitio de unión entre el agonista y el GPCR. Muchos fármacos modernos son antagonistas, como los β-bloqueantes utilizados en el tratamiento de trastornos relacionados con el corazón y la hipertensión (el Premio Nobel de Medicina de 1988 fue obtenido por Sir James W. Black por descubrir el propranolol, que bloquea el receptor adrenérgico β de la adrenalina). Otros son agonistas que activan, por ejemplo, los receptores de la dopamina y la serotonina para aliviar la enfermedad de Parkinson, migrañas y trastornos neuropsiquiátricos, o son agonistas inversos, que impiden que la actividad basal de, por ejemplo, el receptor GABA involucrado en la memoria y el aprendizaje. Las aplicaciones farmacológicas de los trabajos de los ganadores del Premio Nobel de Química de 2012 son realmente incontables.
Los interesados en más detalles, de primera mano, disfrutarán con estas dos charlas en youtube del propio Lefkowitz en las que nos relata su descubrimiento de los GPCR. Aunque están en inglés, realmente merecen la pena.
XVII Carnaval Biología: Nuevo avance en la biofísicoquímica de la fotosíntesis en las plantas

Lo primero, te recomiendo ver el Discurshow «Protón» de Xurxo Mariño y Vicente de Souza, aunque no mencionen la fotosíntesis, en la que el protón tiene un papel fundamental. La fotosíntesis transforma la luz del Sol en energía química a partir de dos moléculas de agua, que se descomponen en una molécula de oxígeno O2, junto a cuatro protones (núcleos de hidrógeno o iones H+) y cuatro electrones. La fotosíntesis en cianobacterias, algas y plantas se denomina fotosíntesis oxigénica y se basa en el llamado sistema fotosintético tipo-II, o fotosistema II (PSII); la secuencia de pasos de esta reacción bioquímica se llama ciclo de Kok (1970) y está catalizada por un complejo Mn4Ca, formado por cuatro átomos de manganeso y uno de calcio. Gracias a las técnicas de espectroscopia se conoce bastante bien su funcionamiento en las escalas de picosegundos, con algunos detalles incluso en la escala de femtosegundos. La reacción química global es 4 YZ(ox) + 2 H2O → 4 YZ(red) + 4 H+ + O2, donde la absorción de fotones con una longitud de onda alrededor de 680 nm oxida cuatro moléculas de tirosina, YZ(ox), que actúan a su vez como oxidantes de cuatro moléculas de agua; en este proceso 4 electrones del complejo Mn4Ca se transfieren a las cuatro tirosinas, resultando cuatro YZ(red), mientras que cuatro protones H+ son eliminados del complejo MMn4Ca mediante un proceso de desprotonización (la figura de arriba, parte derecha, muestra los pasos del ciclo de Kok y sus escalas de tiempo). Un nuevo artículo publicado en PNAS ha estudiado en detalle el proceso de transferencia de electrones y protones, rellenando algunos huecos en nuestro conocimiento de esta interesante reacción bioquímica. Hace poco un lector de este blog me pedía que le describiera los detalles cuánticos de la fotosíntesis. Viendo las figuras de esta entrada se puede ver que para entenderlos hay que ser un experto en biofísicoquímica cuántica y yo no lo soy. Aún así, los expertos disfrutarán el artículo de André Klauss, Michael Haumann, Holger Dau, «Alternating electron and proton transfer steps in photosynthetic water oxidation,» PNAS 109: 16035-16040, October 2, 2012.

Entrar en detalles técnicos sería meterme en camisa en once varas, pero quizás conviene poner un ejemplo del nivel de detalle con el que conocemos estas reacciones químicas. Por ello, incluyo aquí esta figura que muestra uno de los pasos del ciclo clásico de Kok de oxidación fotosintética del agua, el paso S2 → S3. Se muestra el complejo Mn4CaO5, la tirosina con actividad redox (Tyr161) y los grupos más importantes que rodean a los enlaces de hidrógeno en esta reacción. Los aminoácidos que se destacan forman parte de la subunidad D1 del PSII, con la excepción e CP43–Arg357. Las moléculas de agua se indican con esferas rojas, los enlaces de hidrógeno con línea a trazos y los protones son esferas grises. La retícula tridimensional en gris representa el complejo de moléculas de agua que incluye 4 HxO en la primera esfera de coordinación del manganeso (Mn4), así como el calcio (Ca) y las tres moléculas de agua de la segunda esfera de coordinación. El primer paso en esta reacción («1st» en la figura) ocurre cuando han pasado menos de 100 ns tras la absorción del fotón y la oxidación de la clorofila primaria del PSII (P680); en este paso Tyr161 (YZ) es oxidada por P680+. La formación de YZ(ox) produce un reordenamiento de la red de enlaces de hidrógeno (que se completa en menos de 1 µs), conduciendo a la transferencia de un protón a His190, desprotonizando una molécula de agua en el complejo mostrado en la retícula tridimensional gris. En el segundo paso de esta reacción («2nd» en la figura), el complejo Mn/YZ pierde un protón en alrededor de 30 µs y se produce la vacante de un protón en el complejo de agua. En el tercer y último paso («3rd» en la figura), en alrededor de 300 µs, la oxidación del manganeso se acopla al paso de transferencia de un protón que ha creado una vacante en el complejo del agua.

Los mecanismos moleculares de las reacciones (foto)químicas del fotosistema II (PSII) se ha estudiado desde los años setenta y se conocen con bastante detalle. La estructura del PSII se determinó por difracción de rayos-X en el año 2004 y contiene unas 20 subunidades proteicas, conocidas como PsbA-Z, en función del nombre de los genes que las codifican, además de un conjunto de cofactores como son las clorofilas (Chl), feofitinas (Phe), carotenoides, hierro, plastoquinonas, complejo de iones Mn (Mn4) y los iones Ca, Cl y HCO. La masa molecular aproximada del PSII es de 320 kDa. Los interesados en más detalles técnicos (en español) pueden recurrir, por ejemplo, al capítulo 2 de la tesis doctoral de Mónica Balsera Diéguez, «Análisis estructural de la proteína extrínseca PsbQ del fotosistema II de plantas superiores,» Universidad de Salamanca, 2004.
Esta entrada participa en el XVII Carnaval de Biología, organizado este mes por el blog «Pero esa es otra historia y debe ser contada en otra ocasión,» cuyo autor @Ununcuadio es buen seguidor de mi blog.
Un español es primer autor de un artículo en Nature que describe cómo actúa cierta proteína transmembranal
Siempre gusta ver a un español de primer autor de un artículo de Nature, aunque F.-Xabier Contreras está afiliado al Centro de Bioquímica de la Universidad de Heidelberg, Alemania. Su artículo presenta un nuevo mecanismo para explicar como una proteína transmembranal (que atraviesa cierta membrana y controla el transporte a través de ella) se activa o desactiva conforme interacciona con ciertos lípidos (esfingolípidos); su descubrimiento se basa en simulaciones por ordenador de dinámica molecular. El vídeo de youtube que abre esta entrada muestra un par de estas simulaciones. No tengo conocimientos suficientes para entrar en detalles técnicos, pero me ha sorprendido el «baile de San Vito» de estas proteínas; uno siempre se imagina que las proteínas son substancias bastante rígidas, pero el vídeo muestra varios grupos funcionales que realizan rotaciones de hasta 360º, como si tuvieran una rótula. Realmente espectacular. El artículo técnico es F.-Xabier Contreras et al., «Molecular recognition of a single sphingolipid species by a protein’s transmembrane domain,» Nature, Published online 09 January 2012.
No tengo conocimientos suficientes para explicar los detalles técnicos del descubrimiento de Xabi, aún así, permíteme unas breves líneas. Las células eucariotas (con núcleo) están formadas por múltiples orgánulos cada con su propia membrana. Hay moléculas que se transportan de unos orgánulos a otros mediante el llamado transporte vesicular. Una serie de marcadores moleculares guían este transporte en cada vesícula como si fueran guardias de tráfico que determinan el orgánulo origen y el destino de cada molécula, así como la dirección del transporte a través de la membrana, si es hacia dentro o hacia afuera de la vesícula. Los esfingolípidos son componentes estructurales de las membranas que pueden actuar como mensajeros intracelulares. No se conoce el mecanismo exacto por el cual las proteínas transmembranales que se encuentran en la bicapa fosfolipídica de cada membrana se activan o desactivan. Xabi y sus colegas han estudiado la interacción entre una proteína transmembranal concreta, llamada p24, y un esfingolípido concreto, llamado esfingomielina SM18. Por lo que parece la proteína presenta dos estados, uno inactivo y otro activo, que se activan por interacción con el esfingolípido, que actúa como mensajero molecular. En este sentido, el esfingolípido actúa como cofactor para la regulación de la función de esta proteína transmembranal. El artículo técnico describe en detalle los cambios en la estructura de la proteína debidos a la interacción con el esfingolípido, detalles estructurales que demuestran la alta especificidad de esta interacción (que como nos aclara @Acebron en los comentarios «esta interacción específica entre SM18 y la proteína es necesaria para la correcta distribución de las vesículas de transporte en las que dicha proteína participa»). Los autores creen que mecanismos similares son responsables de las interacciones entre otros mensajes y otras proteínas transmembranales.
Espero no haber metido mucho la pata. Esta entrada está dedicada a Sergio Pérez Acebron (@Acebron), amigo de Xabi y autor del blog Tall & Cute, quien me retó con un contundente «es una historia muy compleja para divulgar.» No sé si lo he logrado, pero espero al menos haber picado la curiosidad de los biólogos y bioquímicos que lean esto.



